如何量化与模拟微生物群落标准偏差:
筛选计算标准化分数
NGS-based微生物领域的分析是一个转折点,在关键意见领袖和全球联盟呼吁普遍使用定义良好的微生物群落的标准来提高数据精度,允许cross-study和inter-lab比较1 - 5。商用模拟微生物群落的发展有助于增加使用适当的控制,但直到现在,没有简单的方法量化偏见的存在,当这些标准使用。测量完整性系数(筛选)简化了偏差量化一个容易解释,标准化分数和报告。
模拟微生物群是什么?
模拟微生物群落的标准是已知的合成混合微生物物种与一个特定的和准确的百分比。理想情况下,物种为标准选择代表不同的特征,目前微生物研究中可能面临的各种挑战。这些特征包括一系列在细胞壁的韧性和细胞大小细胞溶菌作用偏见挑战,GC含量的光谱测试测序和生物信息学的偏见,和multi-kingdom表示评估物种在不同的测序技术检测。这些光谱允许精度评估微生物工作流,即使标准不包含特定菌株的兴趣。林恩Schriml马里兰大学的研究所基因组科学解释道:“你基准分析是做什么,未必在社区是什么。”6
如何模拟微生物群落控制偏见?
模拟微生物群落标准的目的是作为一个积极的控制,作为一个独特的运行示例在平行研究样本。理想情况下,这种积极的控制是包含在每一个核酸提取发现任何偏差裂解和纯化步骤。如果观察到的相对丰度的标准并不像预期,偏见中引入工作流可能造成标准的转变和未知样品中。现在许多研究采用这一策略包括几个高调的出版物7号到9号。
偏见与一个简单的量化评分,进行筛选
虽然这个领域开始采用适当的控制,彻底不存在简单的解决方案,定量分析的准确性。这个问题已经被提到关键意见领袖,是一个共同讨论在2019年NIST研讨会微生物组的测量标准10。现在,筛选评分系统解决这些问题通过分析微生物标准容易,完全可再生的,和主题分级方案敏感微生物制备方法甚至更小的偏差。
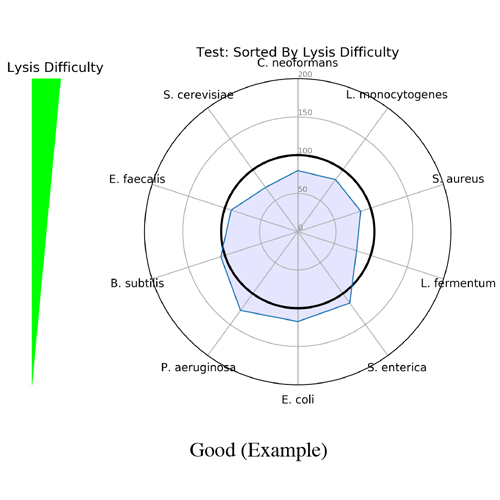
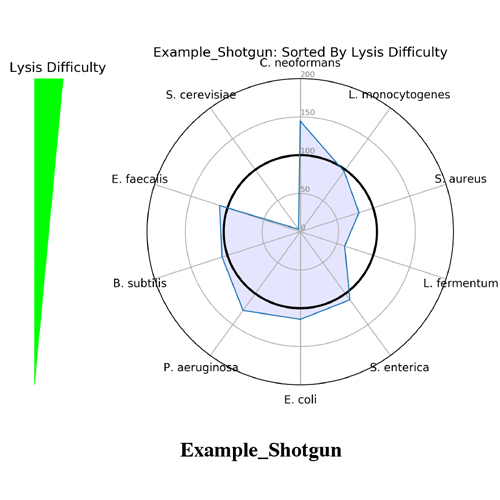
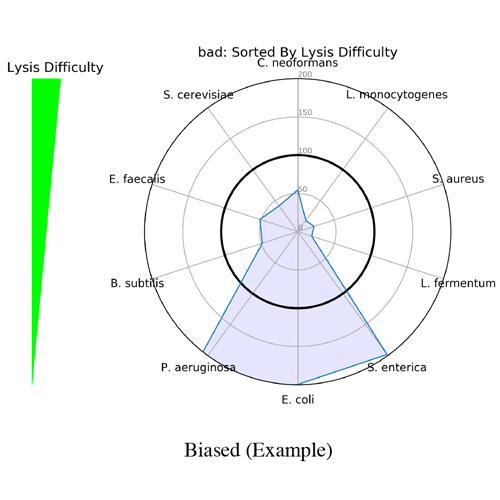
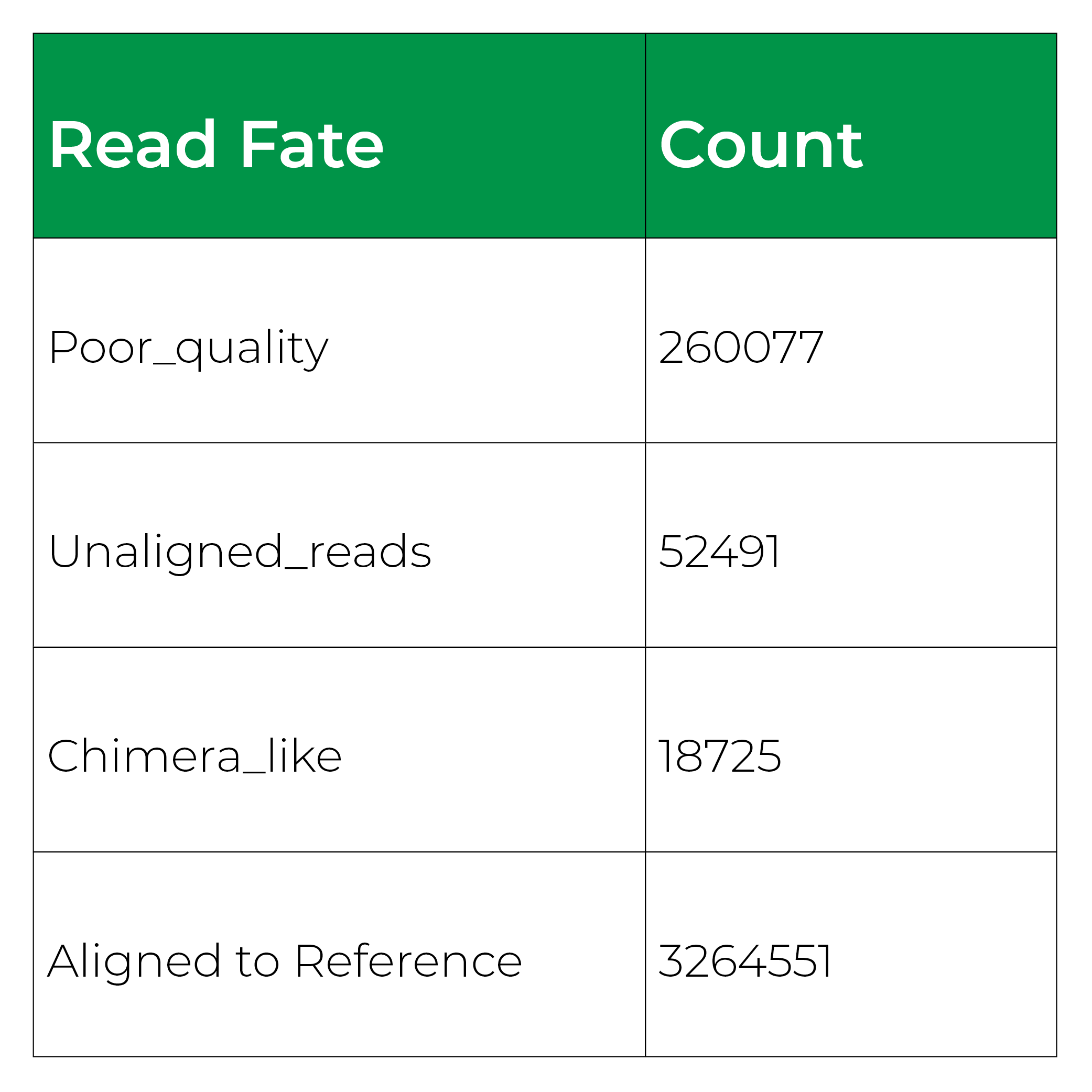
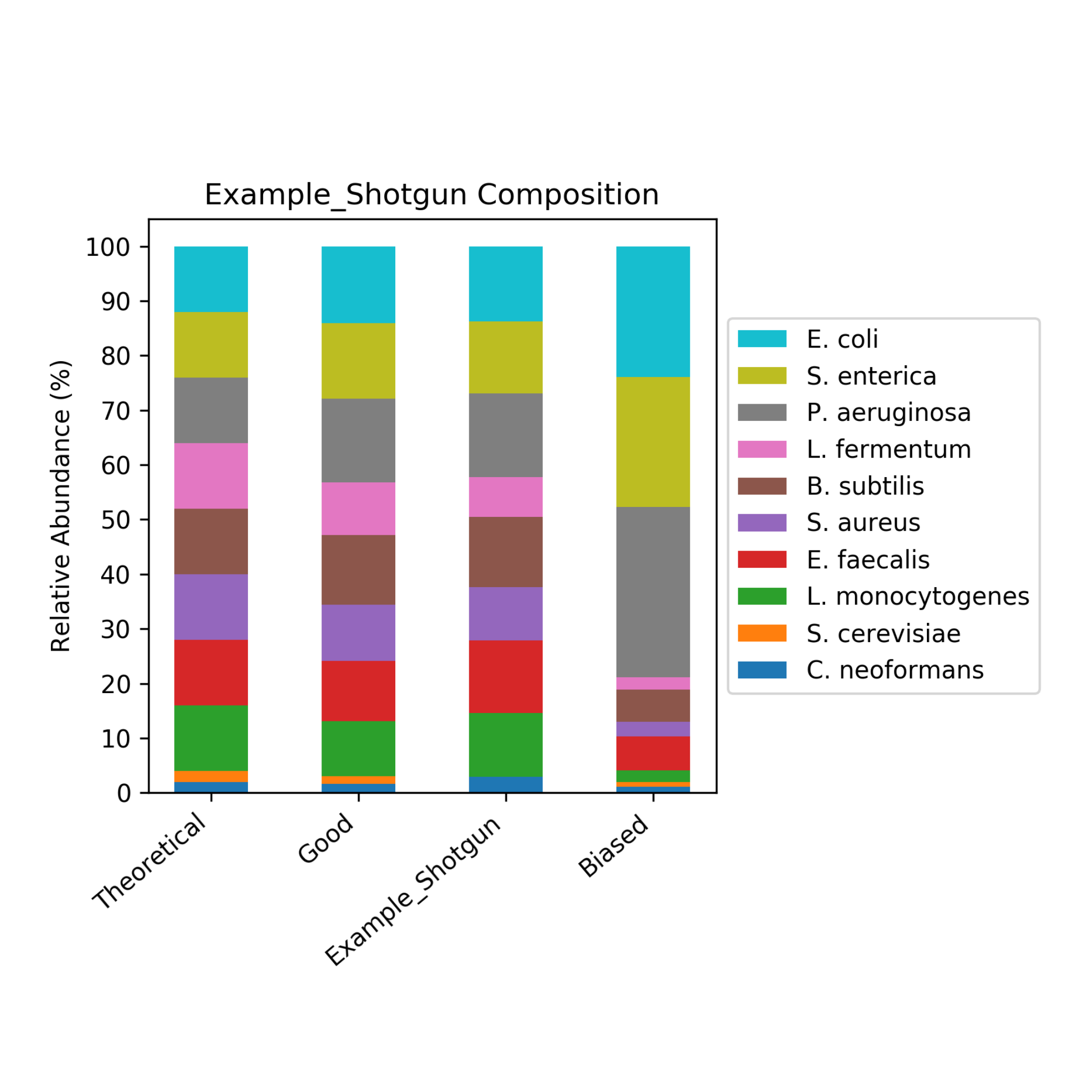
开始作为一个笑话的名字创建它的应用程序后,科学家(迈克)但是,原来测量完整性商完全描述了分数的措施。筛选分数分配的简化了偏见评价分数从0到100,可以解释像高中一年级考试,> 90优秀,80 - 89是好,等等(图1)。这是通过测量测量的忠诚相对丰度相对于一个已知的输入。筛选分数提出了一个用户友好的报告也包括视觉代表评价不同的情节。这些包括雷达情节,类群酒吧的阴谋,和阅读命运数(图2)。
筛选分数是如何计算的?
第一步在评估偏差使用标准模拟微生物群落是理解所使用的制造公差标准。制造公差是一种远离其预期的潜在变化丰富。例如,ZymoBIOMICS微生物群落的制造公差标准是15%,这意味着如果100细胞的目的枯草芽孢杆菌细胞,实际的细胞计数枯草芽孢杆菌可能的范围从8.5到11.5的细胞。重要的是使用一个标准的制造公差,如最小ZymoBIOMICS微生物群落的标准,因为测量的精度评估只是一样好控制。
筛选分数措施偏离预期的相对丰度不能解释为制造公差。它首先分配100%的比例预计每个物种的标准。使用ZymoBIOMICS微生物群落的制造公差标准,任何观察到的相对丰度,超出15%的预计百分比偏差,不能解释为制造公差(图3)。因此,它的偏差可以认为是由一个或多个工作流的偏见造成的。另一方面,如果观察到大量制造公差带内,它可能没有经历任何偏见,会导致偏差(图3)。
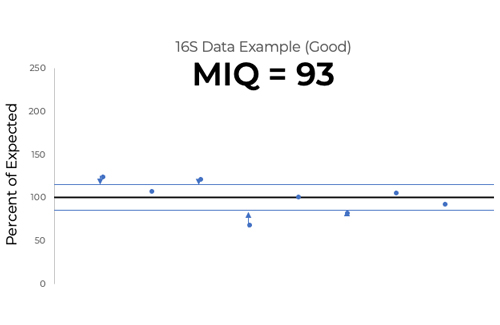
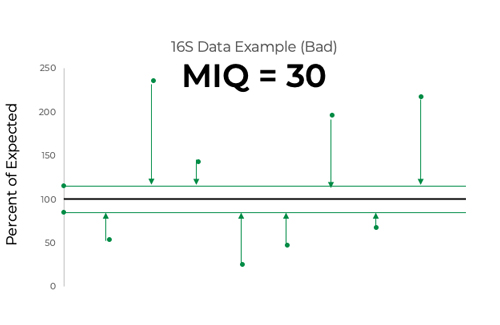
筛选分数分配通过测量的均方根误差(RMSE)观察到的丰度,超出制造公差的乐队。RMSE是一种常用的统计工具用来测量精度相对于一个已知的数量。这些rms减去从100年创建筛选得分。
努力提高微生物的研究
Zymo研究努力的一部分,提高数据质量和再现性微生物,筛选分数应用程序是免费提供给所有人员。版本为16 s和猎枪从Zymo研究数据可以下载源代码的GitHub页面或预先构建的码头工人图像。
今天分析模拟社区数据进行筛选的分数
去进行筛选分数的应用引用
- Sinha R, Abnet CC、白啊,骑士R, Huttenhower C:微生物质量控制项目:基线研究设计和未来的方向。基因组医学杂志2015年,16:276
- Costeaπ,希尔德布兰德F, Arumugam M, Backhed F,布莱塞M,布什曼FD, de Vos WM,埃利希SD,弗雷泽厘米,服部年宏M等:菌群在肠道微生物群落组成的景观。2018年自然微生物,分裂到8 - 16个。3 (1)
- Stulberg E,傅瑞威尔D,普氏LM,默里DM, LoTempio J, Chrisey L,花环J,古德温K, Graber J,哈里斯MC等:美国微生物研究的评估。2016年自然微生物学,1:15015
- 杰克逊SA Kralj詹,林新泽西。NIST /安全部/ FDA报告车间:病原体检测标准生物监控和临床应用。NIST.gov 2018年,特殊(NIST SP) - 1222年出版
- 琼斯MB,汉兰达SK,安德森EL,李W,德瑞M, Klitgord N, Fabani MM, Seguritan V,绿色J,骄傲DT。库制备方法可以影响人类微生物组研究的基因组和功能预测。PNAS上2015年,112年(45):14024 - 14029
- Zymo研究。从直觉到黄金标准。2018。
- 丹科D, et al。全球城市微生物宏基因组地图和抗菌素耐药性。单元2021、184 (13):3376 - 3393。
- 科克加德岩溶年代,ziel4 R, R, Sørensen E,麦当劳D,朱Q, R,骑士阿尔伯特森m .高精度读扩增子序列利用独特的分子标识符与纳米孔或PacBio测序。自然方法2021年18:165 - 169。
- Bullman年代,Pedamallu CS, Sicinska E,克兰西TE,张X, Cai D, Neuberg D,黄K,格瓦拉F,纳尔逊T, Chipashvili O,梭菌属等。分析结直肠癌持久性和抗生素的反应。科学2017,358 (6369):1443 - 1148
- 2019 NIST车间微生物标准测量